
circRNA生产过程包括:模板制备、体外转录、DNA模板去除、环化和纯化。在RNA环化后需要用核糖核酸酶R (Ribonuclease R, RNase R)对未环化的RNA进行消化,达到富集circRNA的目的。当前主流使用的RNase R主要为进口品牌,存在价格高、货期久等问题,不适合用于大批量生产。
翌圣拥有ZymeEditor酶改造平台和专门为GMP级别产品打造的分子酶生产基地,研发生产出纯度高、消化活性强的GMP级别RNase R产品,为新一代RNA疫苗/药物上市提供助力!
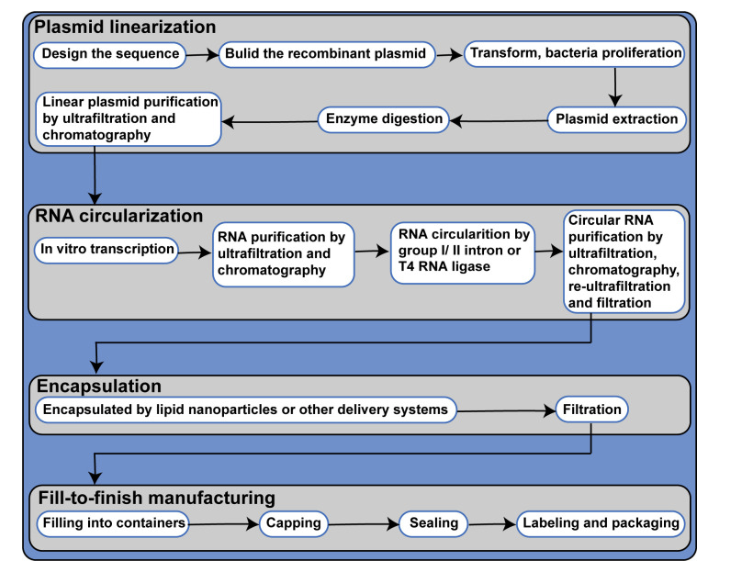
图1.环状RNA 生产流程图
(来源:Front Immunol 2023 Jan 12:13:1091797. doi: 10.3389/fimmu.2022.1091797. eCollection 2022.)
RNase R(Ribonuclease R, RNase R)是一种来源于大肠杆菌的Mg2+依赖性3'→5'核糖核酸外切酶,它能消化所有的线性RNA,不易消化circRNA、套索RNA或3’端突出末端少于7 个核苷酸的双链RNA分子。RNase R常用于去除线性RNA分子,实现富集circRNA和套索RNA等非线性RNA的目的。
主要应用场景为:
1)基因表达研究;
2)可变剪切研究;
3)从生物样本中富集 circRNA;
4)识别内含子套索结构 RNA;
5)鉴定外显子 circRNA。
产品特点
·
蛋白纯度(SDS-PAGE)≥95%;
·
无核酸外切酶残留;
·
与竞品相比,产品消化线性RNA能力相当;
·
与竞品相比,产品对circRNA富集作用相当。
应用案例
案例一
与A进口品牌RNase R产品消化线性RNA能力相当
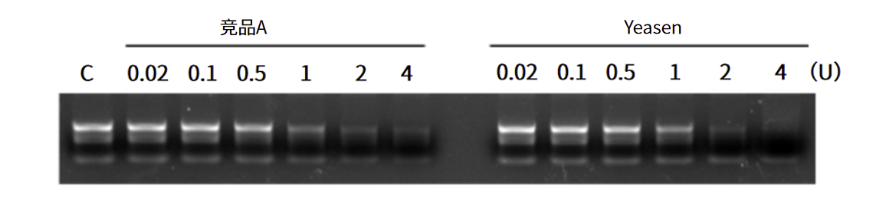
图.翌圣RNase R产品与A进口厂家RNase R产品消化线性RNA能力比较:将翌圣RNase R产品与A进口厂家RNase R产品分别投入含有线性RNA底物的反应体系中进行反应,结果显示当RNase R产品的投入量达到2-4 U时,可观察到RNA条带变微弱甚至消失,表明RNase R对线性RNA分子具有消化作用,且翌圣RNase R产品与A进口厂家RNase R产品对线性RNA分子的消化能力相当。
与A进口品牌RNase R产品对circRNA富集作用相当
案例二
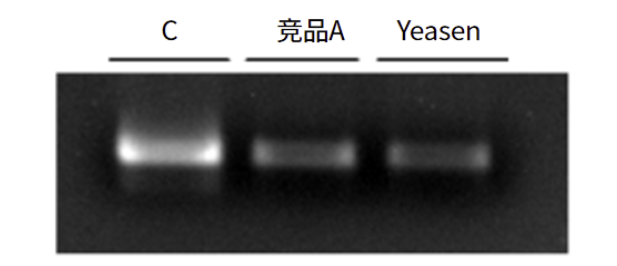
图.翌圣RNase R产品与A进口厂家RNase R产品对circRNA的富集作用比较:将翌圣产品与A进口厂家RNase R产品分别投入含有circRNA底物的反应体系中进行反应,结果显示经过翌圣RNase R产品与A进口厂家RNase R产品处理后的RNA条带亮度基本无差别,表明circRNA能耐受RNase R的消化。以上结果证实翌圣产品与A进口厂家RNase R产品均能有效用于circRNA富集。
案例三
翌圣RNase R对线性RNA的切割效果优于其他品牌
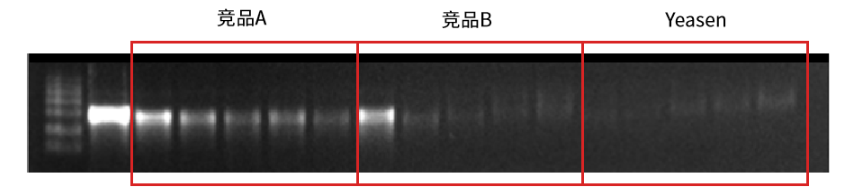
图.翌圣产品与厂家A和B的RNase R消化线性mRNA分子效果比较:将翌圣RNase R产品与厂家A和B的RNase R产品分别投入含有1 μg 线性mRNA底物的反应体系中进行反应,结果显示翌圣RNase R产品对线性RNA的消化效果优于厂家A和B的RNase R。
产品性能
蛋白纯度(SDS-PAGE)≥95%
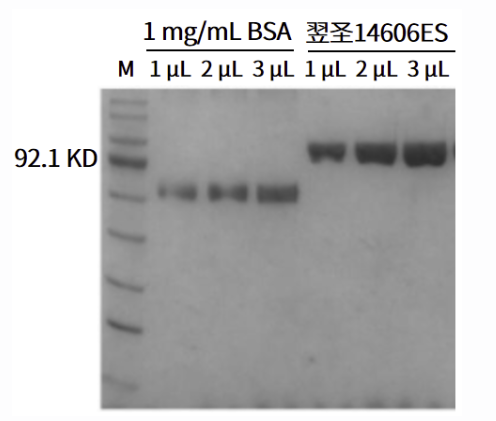
图.蛋白纯度检测:使用SDS-PAGE技术检测产品的蛋白纯度,从左到右依次为相同产品浓度下不同体积(1 μL,2 μL,3 μL)的结果,结果表明翌圣RNase R产品蛋白纯度≥95%。
无核酸外切酶残留
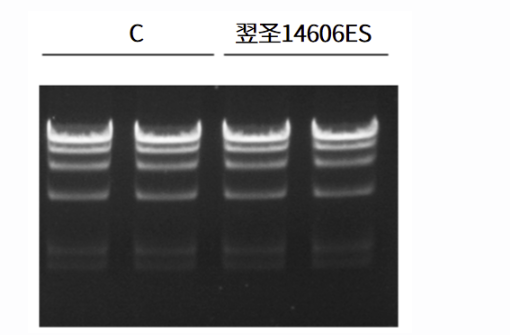
图.核酸外切酶残留检测:将20 U 翌圣RNase R加入到 0.5 μg λDNA- Hind Ⅲ digest中,于37℃孵育4小时,之后进行琼脂糖凝胶电泳 ,结果显示DNA 条带未发生变化,说明翌圣RNase R产品无核酸外切酶残留。
翌圣环状RNA 解决方案
产品信息
|
应用场景 |
产品名称 |
产品货号 |
|
模板制备 |
10664ES |
|
|
10661ES |
||
|
体外转录 |
10623ES |
|
|
10629ES |
||
|
10625ES |
||
|
10620ES |
||
|
10621ES |
||
|
10670ES |
||
|
10652ES |
||
|
10653ES |
||
|
10654ES |
||
|
10655ES |
||
|
环化 |
14652ES |
|
|
14651ES |
||
|
纯化 |
14606ES |
|
|
14615ES |
||
|
14616ES |
参考文献:
1. Qu L, Yi Z, Shen Y, et al. Circular RNA vaccines against SARS-CoV-2 and emerging variants. Cell. 2022;185(10):1728-1744.e16. doi:10.1016/j.cell.2022.03.044
2. Chen L, Wang C, Sun H, et al. The bioinformatics toolbox for circRNA discovery and analysis. Brief Bioinform. 2021;22(2):1706-1728. doi:10.1093/bib/bbaa001
